搜索简单基因组中的重复序列
文章目录
分析原核/病毒基因组中的重复序列
寻找基因间区
首先,计算基因组中的基因编码区。然后,通过interval取反得到基因间区。若只为得到基因间区,重叠的CDS区域不用合并,并不影响与基因组全长取差。
|
|
检测简单重复序列
TRF, REPuter是常用的检测简单重复序列的工具,可在全基因组范围内寻找,也可针对特定基因间区进行搜索。
TRF一般用于寻找串联重复序列,可使用在线服务。
REPuter不仅可寻找串联重复,对于4种类型的重复序列(如下)均可检索。使用比勒费尔德大学生信中心提供的在线服务,结果可通过网页可视化。
- forward(direct) match

- reverse match
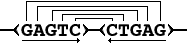
- complement match
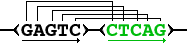
- palindromic match

blast2seq 是NCBI较为古老的BLAST工具中的一员。可直接对两段序列进行比较,不需要make database. 对同一段序列进行自比较,并过滤去同位置完全匹配的结果,即可用于发现其中存在的重复序列。
检测回文序列
EMBOSS palindrome可检测序列内部的回文序列。通过参数可设定回文序列的长度范围,错配的碱基数。
