如何绘制一张热图
文章目录
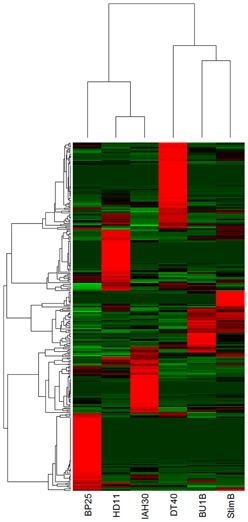
绘制热图的过程分为两个关键的步骤,第一,将数据聚类;第二,将数值矩阵转化为颜色图像。
将数据聚类
聚类的方法有许多,其中最简单的方法就是分层聚集聚类(hierarchical, agglomerative cluster analysis).
简单来说,该方法会首先计算矩阵中所有数据点之间的相互距离,然后将相聚最近的数据点连接起来,然后连接距离第二近的数据点,重复直至所有数据点都被聚类。我们得到的聚类树就是这一过程的展示。
聚类过程中,有时需要将不同的类再次聚类,这个过程也有许多不同的方法,但是最常用的方式是计算两个类中所有点之间的平均距离。
计算距离
距离代表着两个数据点之间的差异。这与相似性测量是正交的(相互独立的)。
距离是如何计算的呢?在R的heatmap()或heatmap.2()函数的默认方法是使用dist()函数。dist()函数的默认方式是欧几里得距离。这一方法测量的是数据向量之间的绝对距离,但是却与其表达模式( the “shape” of the “curve”)无关。
例如,测定4个基因在8个时间点的表达量。
|
|

可以看到,图中表达的基因与两个低表达的基因。关键的是,这两对基因的表达模式完全相反。
如果我们将这个数据置于dist()函数中,可得到这样的距离矩阵
|
|
|
|
距离矩阵中最小的值是l1与l2之间的距离(5.65),因此这两者被聚为一类;然后,次小值是h1与h2之间的距离(28.28),因此,这两者接下来被聚类;最后,这两个类连接起来。这就产生了一个基于距离矩阵的简单聚类分析。
|
|

简单的热图(列水平上不聚类,保持时间点的顺序)
|
|

聚类过程按照距离进行,l1与l2是最相似的,因此它们被聚为一类;然后,h1与h2被聚类。但是,由于颜色不合适,热图看起来很糟糕。尽管l1与l2被聚为一类,它们的颜色却不是同一个模式;h1与h2也是如此。此外,l1与h1有相同的颜色,尽管它们的表达量相差很大。
数据缩放(scaling)
l2与h2有着相同的颜色,但实际上它们的表达量不同。这是因为它们的表达量被缩放了,然后再转化为热图中的颜色。heatmap()与heatmap.2()中的默认设置都是按照行缩放(scale by row). 数据矩阵按行依次使用scale()函数进行缩放,然后转换为颜色。
关闭缩放后,我们可以看到热图通过欧几里得距离聚类后的结果。
|
|

这样看起来结果好了一些,但是还不完善。l1与l2都是绿色,而h1与h2都是红色/黑色。
一般来说,热图中的绿色代表较低的数值;红色代表较高的数值;黑色则代表中间值。在不进行缩放的时候,l1与l2都是较低的值,因此,它们是绿色;h1与h2中的较大值是较高的值,因此为红色;其中较低的值则是中间值,因此为黑色。
问题所在
通常情况下,我们系统将有相近表达模式的基因聚类在一起。而有些基因的表达量高,有些基因的表达量低。将所有高表达的基因标为红色,低表达的基因表为绿色,而不考虑随时间表达模式的变化。有些时候,我们希望这样,但更多时候,我们系统将相同表达模式的基因聚类在一起。
因此,heatmap()和heatmap.2()的默认参数结果对我们不可用。它们默认都缩放了数据,这在所有数据点都有着相近表达模式时很有用,但是它们还是用了欧几里得距离,这在我们系统通过表达模式聚类时并不适用。
我们知道h1与l1有相近的表达模式(similar shape),h2与l2也有相近的表达模式。但是,dist()函数只考虑绝对距离,并不考虑其表达模式。
解决方案
使用另一种距离计算方式,相似性计算,皮尔逊相关系数(the pearson correlation co-efficient)。简单来说,它产生一个在-1到1之间的值;1代表两者相同;-1则表达两者正好相对。
其计算结果如下:
|
|
|
|
结果中,h1与h2的相关系数为-1, 因此它们非常不相似,而h1与l1,h2与l2则完全正相关。
在聚类过程中,我们需要的时距离矩阵,不是相似性矩阵。因此,我们需要用1减去相似性,转换得到:
|
|
|
|
这样h1与l1之间的距离为0;h1与h2之间的距离为2. 那么我们可以得到简单的聚类结果:
|
|

简单的热图结果
|
|

如此,相同表达模式的基因被聚类在一起,也具有相同的颜色。
热图实例
来自ARK-Genomic的实例
|
|
因此,热图绘制还是一个比较复杂的可视化过程。
无量纲化
极差法
(观测值-最小值)/极差
将数值缩放到0至1之间,又称为归一化(normalization)
log函数标准化
log10(X)/log10(MAX)
适用于观测值大于等于1的情况,这也是一种特殊的归一化。
z-score
(观测值-均值)/标准差
将数值转化为均值为0,方差为1的标准正态分布的形式,又称作标准化(standrdization)
参考来源
http://www.opiniomics.org/you-probably-dont-understand-heatmaps/
https://sebastianraschka.com/Articles/2014_about_feature_scaling.html

